Benchmarking Universal Single-Copy Orthologs
随着基因组数据越来越多,但是将短reads组装成全长的染色体依然非常具有挑战,因此我们经常要对组装的基因组进行质量评估。进行质量评估时常用的方法有:碱基错误率的统计、插入片段长度统计、k-mer分布、contig长度分布、N50等等。然而这些统计方法并没有从基因含量层面来评估基因组的完整性,而基因含量是可以影响数据解读和下游分析的重要指标。
而BUSCO是用于在基因含量层面来评估基因组完整性的一个公开发表流程,其原理在于:先构建一个Benchmarking Universal Single-Copy Orthologs (BUSCO) 的数据库,这个数据库有很多种,有真核生物的数据库,脊椎动物的数据库,鸟类的数据库。以脊椎动物数据库为例,这个数据库中的所有基因都在脊椎动物中存在且只存在一个拷贝,因此我们就可以以此为标准,对组装的基因组用这些基因集进行注释(这个过程中用到了Augstus),通过注释情况来评估不同基因组的质量。
由于我们是做鸟类基因组研究,而Gallus gallus即原鸡的基因组是鸟类基因组研究中非常重要的模式基因组,而从14年到现在,已经从第4个基因组版本更新到了第6个基因组版本,虽然从N50等指标上来看越新的基因组其质量越好,但是我们还是想看一看是不是从基因含量的层面上版本越新其基因组完整性越好。于是我用了三个数据库(真核生物、原核生物、鸟类)分别对gal4、gal5、gal6的基因含量进行了评估,然后得到了一下统计结果:
真核生物BUSCO数据库: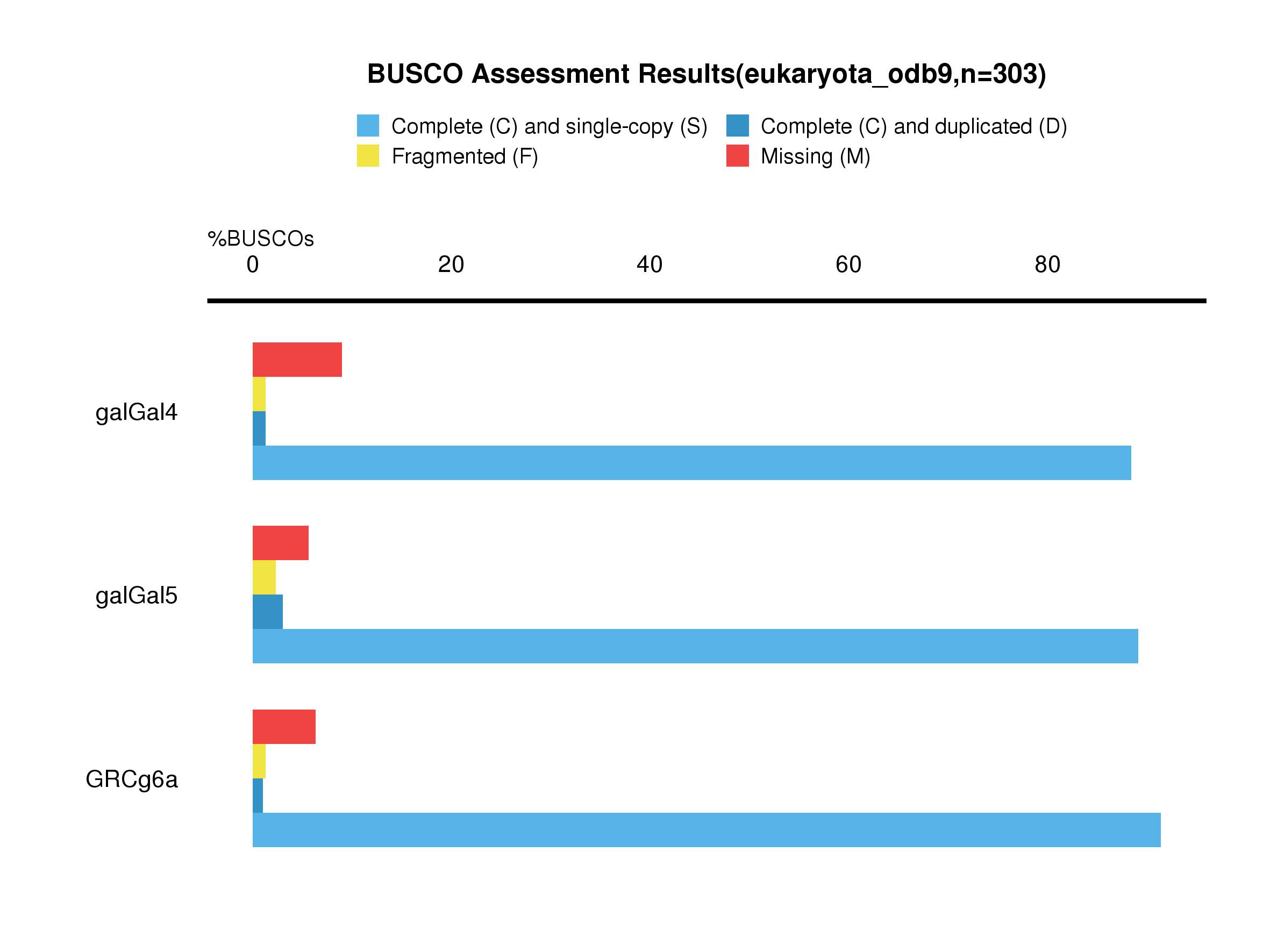
不同指标的含义:
Complete:完整性,在被评估的基因组中BUSCO基因的预测分数以及比对上的长度打到了要求
Complete and single-copy:完整且在该基因组中只有一个拷贝的数目,该指标越高说明组装效果越好
Complete and duplicated:完整且在该基因组中有多个拷贝的数目,由于BUSCO基因集中的基因都是单拷贝的基因,因此如果出现了多个拷贝,就说明在单体型组装过程中出现了错误,因此该指标越高说明组装效果越差
Fragmented:Fragmented的基因虽然达到了预测分数,但是长度并没达到要求。有可能基因组组装过程中没有将该片段组装出来,也有可能是基因预测的时候由于基因结构的特殊性没能完整地将该基因预测出来。该指标越低越好。
Missing:Fragmented的基因在基因组中完全没有找到,或者预测分数低于要求。有可能基因组组装过程中没有将该片段组装出来,也有可能是基因预测的时候由于基因结构的特殊性没能完整地将该基因预测出来。该指标越低越好。
因此从真核生物BUSCO数据库的评价结果来说,Gal6综合情况最好,gal5和gal4之间则难分伯仲。
脊椎动物BUSCO数据库: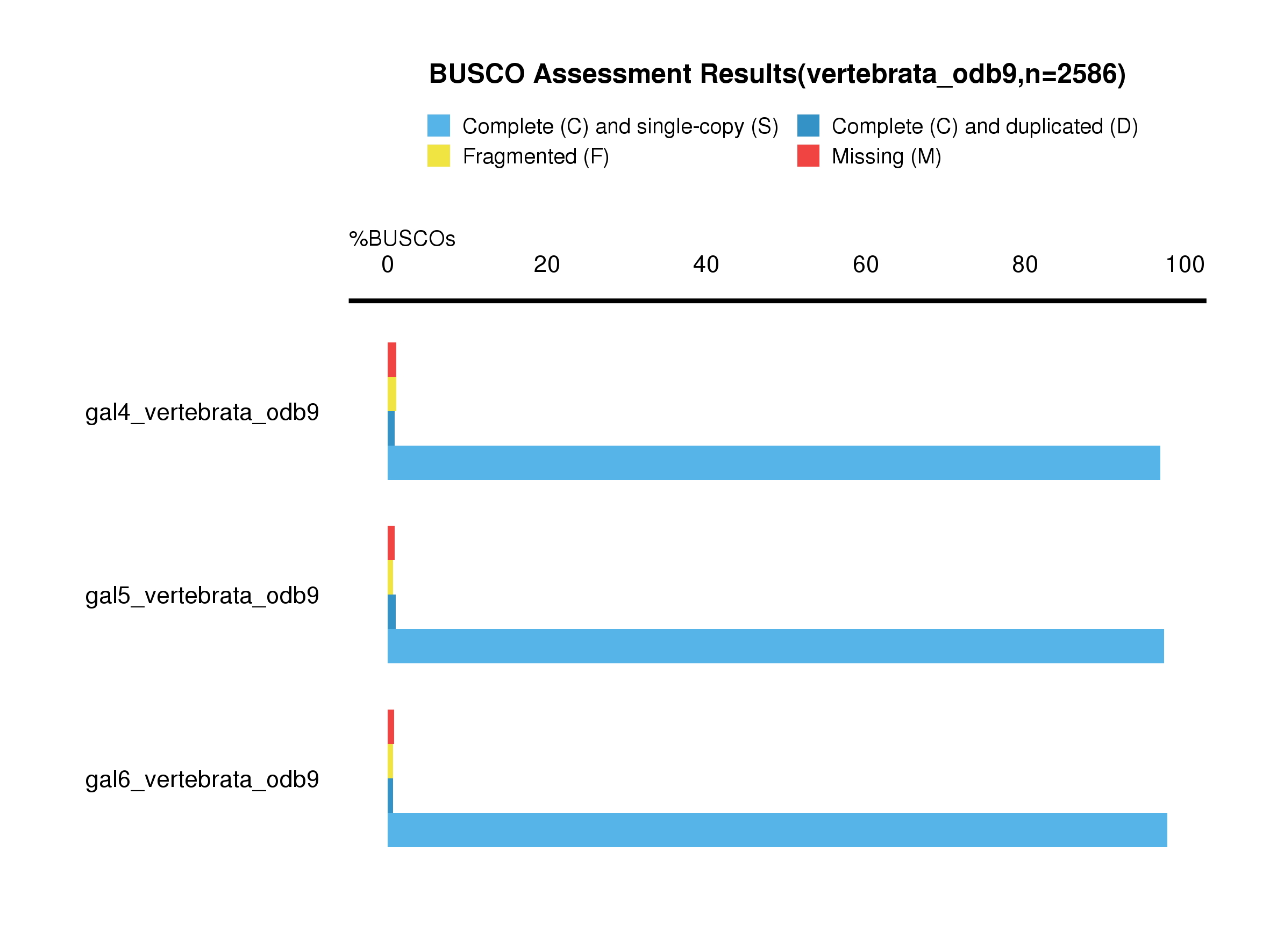
从真核生物BUSCO数据库的评价结果来说,Gal6综合情况最好,gal5次之,gal4最好。
鸟类BUSCO数据库: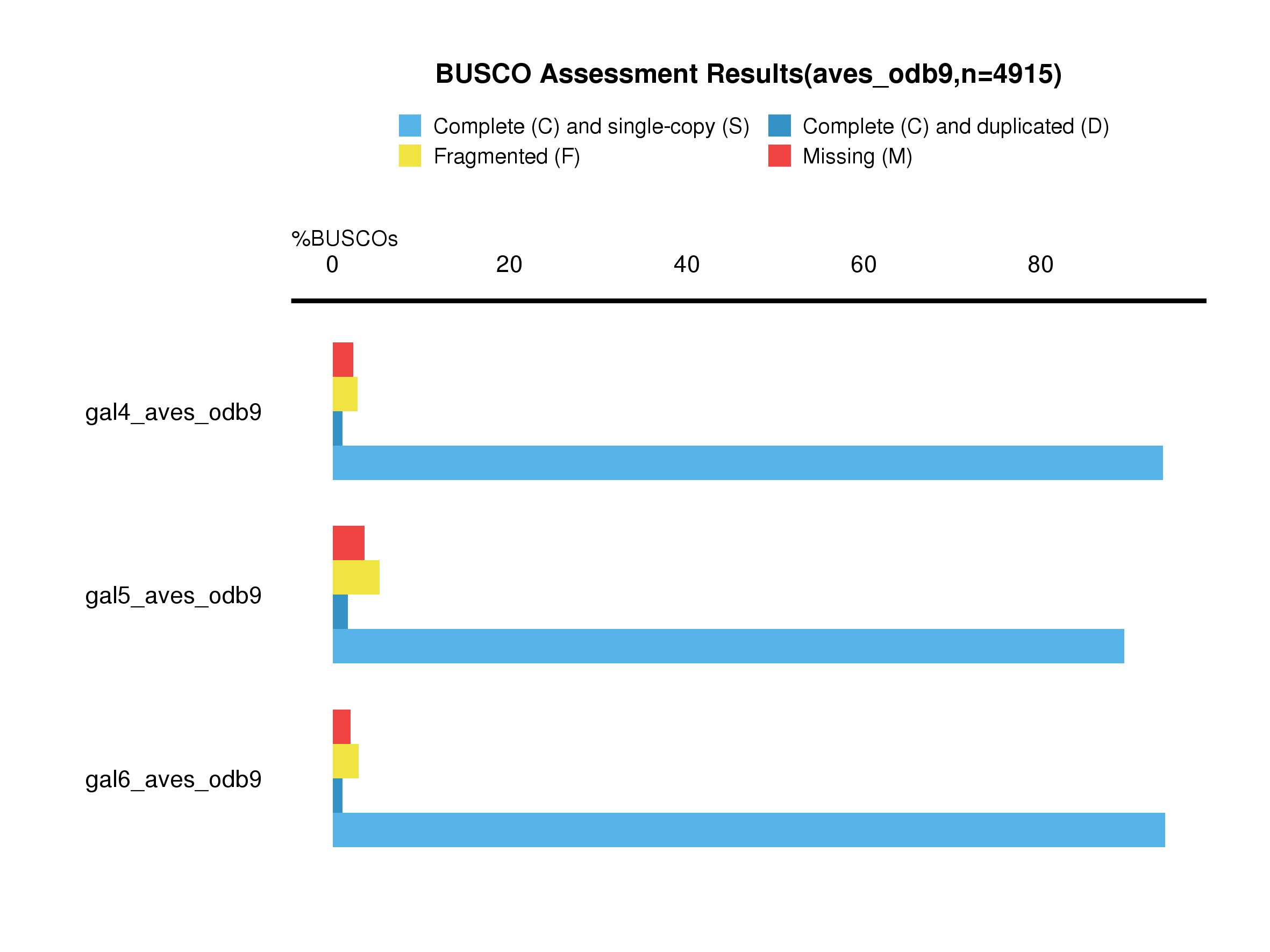
从真核生物BUSCO数据库的评价结果来说,Gal6综合情况最好,gal4次之,gal5最好。
但是根据BUSCO提供的使用说明来看,在评估基因组时应该选取最specific的数据库来进行评价,比如我们在评估鸟类基因组时,就应该以鸟类的数据库为标准而非以脊椎动物的数据库为标准。
综上,Gal6综合情况最好,gal4次之,gal5最次。
BUSCO的流程中有一个绘图脚本可以对BUSCO的结果进行画图,但是其输出结果的画风是这样的: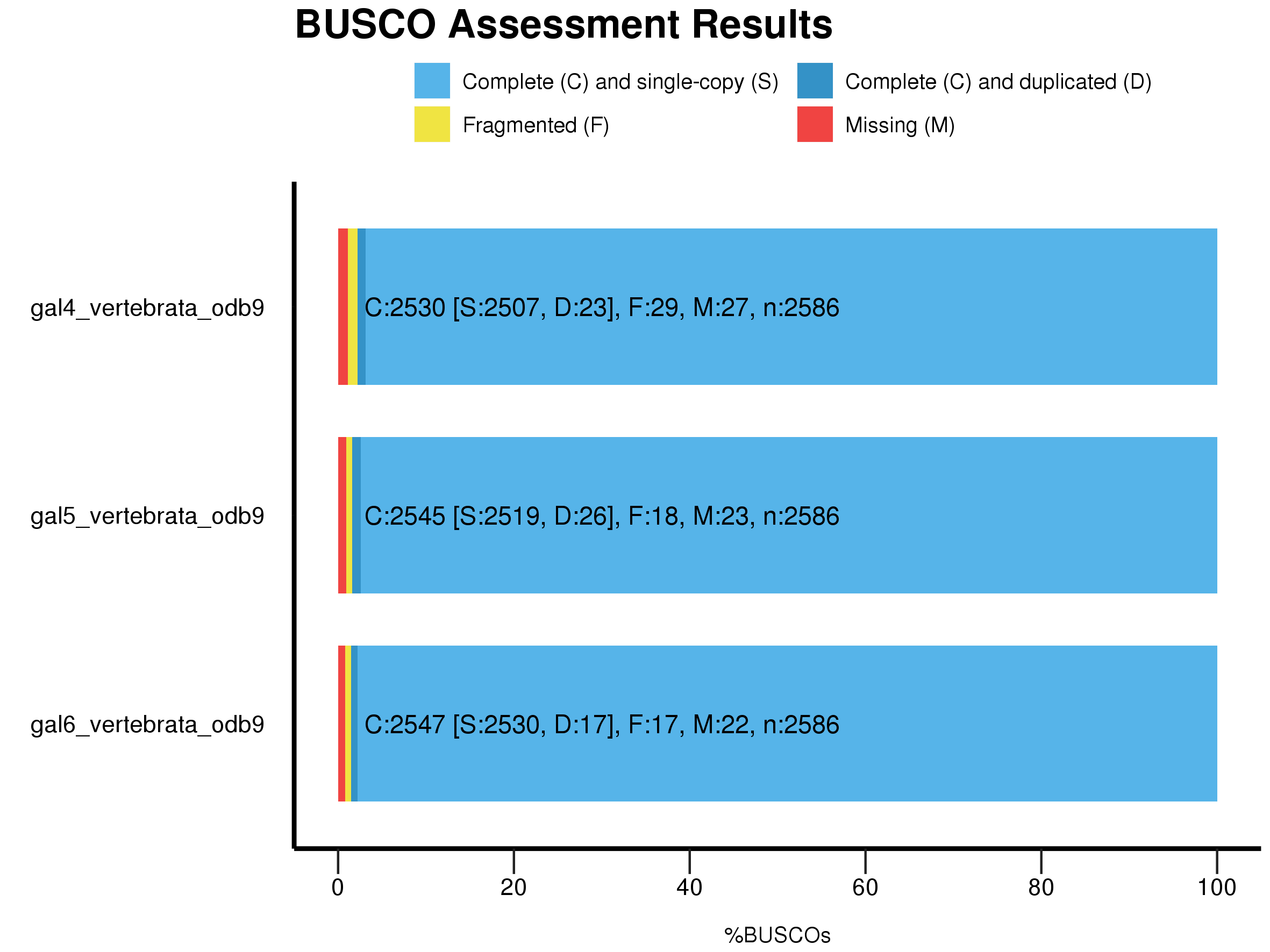
但是根据我之前看的《用数据讲故事》这本书中所提供的绘图原则(推荐下这本书,这本书里面所讲的绘图原则很实用,可以让你知道数据可视化展示时的基本原则,回头我也会总结下这本书的要点,作者博客),该图有三个地方需要改进::
1.堆积条形图不利于体现差别,因此用簇状条形图更好
2.Y轴的坐标轴其实没有也能看清
3.X轴在最上面更符合阅读顺序。
因此我修改了BUSCO自带的绘图脚本,地址:https://github.com/xieduo7/my_script/blob/master/busco_plot/generate_plot_xd_v2.py
参考文献
Simão, F. A., et al. (2015). “BUSCO: assessing genome assembly and annotation completeness with single-copy orthologs.” Bioinformatics 31(19): 3210-3212.
Waterhouse, R. M., et al. (2018). “BUSCO Applications from Quality Assessments to Gene Prediction and Phylogenomics.” Molecular Biology and Evolution 35(3): 543-548.
Korlach, J., et al. (2017). “De novo PacBio long-read and phased avian genome assemblies correct and add to reference genes generated with intermediate and short reads.” GigaScience 6(10): 1-16.